In questi giorni segnati dall’arrivo dell’emergenza Coronavirus in Italia, molti si saranno domandati quali fossero gli strumenti scientifici esistenti per prevedere il propagarsi dell’epidemia e anche se fosse possibile capire se certe misure restrittive fossero realmente utili. A questo proposito è noto che esistono numerosi modelli matematici capaci di descrivere in modo quantitativo il diffondersi di un fenomeno infettivo. Segnaliamo in rete negli ultimi giorni un buon articolo sul Corriere della Sera di Paolo Giordano, un articolo su Plus Magazine di Marianne Freiberger, e due articoli divulgativi di Paolo Alessandrini nel suo blog (parte prima e parte seconda). Inoltre pochi giorni fa la Ilaria Dorigatti, matematica e ricercatrice del Centre for Global Infectious Disease Analysis all’Imperial college di Londra, è stata intervista da Radio3 Scienza a proposito di scenari virali. Qui sul sito vi proponiamo un approfondimento tematico di Andrea Pugliese, professore di analisi matematica presso l’Università di Trento, che è il maggior studioso italiano di modelli matematici per le epidemie.
Il modello SIR di Kermack e McKendrick
Sono quasi cento anni che si usano modelli matematici per descrivere il propagarsi delle epidemie. Infatti i modelli attualmente usati in gran parte originano dal modello proposto da Kermack e McKendrick nel 19271. Anche se il modello contiene ipotesi chiaramente irrealistiche, i concetti introdotti tramite tale modello risultano essenziali per fornire una prima intuizione sulla dinamica delle epidemie, intuizione che rimane confermata in modelli più complessi, sia pure con numerose modifiche.
Tale modello prevede la divisione della popolazione in compartimenti, quello dei suscettibili, S, gli individui sani che possono essere infettati, gli infetti e i rimossi R, compartimento che comprende coloro che sono guariti e immuni e (nella trattazione originale) anche coloro che sono morti per la malattia.
Nella trattazione originale gli infetti vengono distinti a seconda del tempo \tau trascorso dal momento dell’infezione (li chiameremo infetti con età di infezione \tau); poniamo \pi(\tau) la probabilità che un individuo sia ancora infetto \tau tempo dopo l’infezione (in caso contrario sarà diventato un rimosso), e \phi(\tau) la sua infettività (possiamo definirla come la probabilità che un suscettibile contattato da un infetto a quello stadio diventi infetto).
In teoria le funzioni \pi(\tau) e \phi(\tau) potrebbero essere stimate a partire da dati sperimentali per infezioni animali in laboratorio. In pratica, per infezioni umane, a partire dall’analisi di casi in cui si riesca ad accertare il momento del contagio, si stimano tramite metodi statistici due quantità collegate al periodo di infettività: il periodo di incubazione (ossia l’intervallo di tempo fra il momento di infezione e la comparsa di sintomi) e l’intervallo seriale, ossia il tempo fra la comparsa dei sintomi in un infetto e la comparsa dei sintomi in un individuo infettato dal primo. Per esempio per il coronavirus, Li et al.2 hanno stimato, sulla base di pochissimi casi, un tempo medio di incubazione pari a 5,2 giorni e un intervallo seriale medio di 7,5 giorni, in entrambi i casi con intervalli di confidenza molto ampi.
Per completare il modello è necessario stabilire la legge secondo la quale si infettano i suscettibili. L’ipotesi fatta da Kermack e Mckendrick è quella dell’azione di massa ovvero del “mixing” omogeneo; si assume cioè che ogni individuo abbia la stessa probabilità di contattare qualunque altro individuo nella popolazione, indipendentemente dai contatti passati. Si capisce come tale ipotesi sia irrealistica: siamo spesso in contatto con le persone della nostra famiglia, del nostro luogo di lavoro, classe scolastica, gruppo di amici e molto di rado con chi abita in un luogo diverso, ha età e professioni diverse. Negli ultimi anni si sono quindi sviluppati modelli computazionali che cercano di tenere conto di molte caratteristiche degli individui per arrivare a previsioni più accurate; però la semplicità della legge di azione di massa permette di effettuare un’analisi teorica precisa e quindi di raggiungere conclusioni potenti.
Supponiamo allora che ogni individuo, e in particolare ogni suscettibile, contatti in media \beta individui nell’unità di tempo e si vuole modellizzare l’incidenza i(t), ossia il tasso di nuove infezioni; in altre parole il numero di nuove infezioni nell’intervallo infinitesimo (t,t+dt) è dato da i(t)\,dt. Al tempo t gli individui con età di infezione \tau sono i(t-\tau) \pi(\tau): coloro che si sono infettati al tempo t-\tau e sono ancora infetti. La probabilità che un individuo contattato sia un infetto con età di infezione \tau è quindi pari a \frac{i(t-\tau) \pi(\tau)}{ N(t)}, dove N(t) è la popolazione totale; infine \phi(\tau) è la probabilità di infettarsi se si ha contattato un infetto con età di infezione \tau.
Integrando su tutte le possibili età di infezione (che per comodità matematica supponiamo possano essere qualunque numero positivo) otteniamo l’equazione \label{incidence} i(t) = S(t) \beta \int_0^\infty \frac{i(t-\tau) \pi(\tau) \phi(\tau) }{ N(t)}\, d\tau. Per completare il sistema supponiamo che l’epidemia si svolga su un tempo sufficientemente breve da poter ignorare nascite e morti per altre cause, e che anche le morti causate dalla malattia siano sufficientemente poche rispetto alla popolazione totale, da poter supporre che N(t) sia una costante N. Quindi S(t) cambia solamente a causa di nuove infezioni e otteniamo il sistema \label{system1} (1)\ \ \ \ \ \begin{array}{lcl} S'(t) & = & – i(t) \\ i(t) & = & \beta \frac{S(t)}{N } \int_0^\infty i(t-\tau) \pi(\tau) \phi(\tau)\, d\tau. \end{array} La seconda equazione presuppone che l’infezione proceda da un tempo infinito, e ciò porrebbe problemi sia biologici sia matematici. Si può supporre invece che l’infezione inizi al tempo 0 con un certo numero di infetti di diverse età di infezione; modificando il sistema per tenerne conto, si arriva a un sistema accoppiato di un’equazione differenziale e di un’equazione integrale di Volterra, che matematicamente è ben posto.
Crescita esponenziale e numero di riproduzione di base R_0
L’analisi del sistema (1) necessita di strumenti avanzati, ma la conclusione sull’andamento iniziale dell’epidemia è relativamente semplice. All’inizio dell’epidemia gli infetti e le nuove infezioni sono poche e si può quindi approssimare S(t) \approx S_0, ottenendo un’equazione lineare per i(t): \label{incidence_linear} (2) \ \ \ \ \ \ i(t) = \beta u_0\int_0^\infty i(t-\tau) \pi(\tau) \phi(\tau) \, d\tau. dove u_0 = S_0/N. Nel caso di una nuova infezione, non vi è immunità pregressa e quindi S_0 \approx N, ossia u_0 \approx 1. Si dimostra che le soluzioni di (2) crescono (o decrescono) esponenzialmente, i(t) \approx C e^{rt}, dove r è l’unica soluzione reale dell’equazione trascendente \label{trova_r} (3)\ \ \ \ \ \ \ \beta u_0\int_0^\infty \pi(\tau) \phi(\tau) e^{-r \tau} \, d\tau = 1. Si vede facilmente che r > 0 [ovvero r < 0], ossia l’epidemia cresce [ovvero decresce] se e solo se R_0 u_0 > 1 [ovvero R_0 u_0 < 1] dove \label{R0} (4)\ \ \ \ \ \ \ R_0 = \beta \int_0^\infty \pi(\tau) \phi(\tau) \, d\tau. R_0 è detto il numero di riproduzione di base dell’epidemia e rappresenta il numero medio di individui infettati da un infetto nel corso del suo periodo infettivo, supponendo che ogni altro individuo nella popolazione sia suscettibile. Infatti, se pensiamo a un individuo infettato al tempo 0, esso sarà ancora infetto al tempo \tau con probabilità \pi(\tau), a quel tempo contatterà altri individui (necessariamente suscettibili) al tasso \beta e li infetterà con probabilità \phi(\tau). Integrando su tutti i tempi \tau da 0 a \infty, si trova il numero totale diindividui infettati lungo tutto il periodo infettivo. Se invece u_0 è la proporzione di popolazione suscettibile, il numero totale infettato sarà R_0 u_0.
La condizione R_0 u_0 > 1 perché un’epidemia si possa diffondere (il cosiddetto teorema di soglia di Kermack e McKendrick) diventa allora molto intuitiva. Se R_0 u_0 > 1, ogni individuo infetto ne infetta in media più di 1, che a sua volta ne infetterà più di 1, e cosı̀ via, con una reazione a catena esplosiva. Se invece R_0 u_0 < 1, ogni individuo infetto ne infetta in media meno di 1, che a sua volta ne infetterà meno di 1, e quindi l’epidemia si estinguerà velocemente da sola.
L’importanza della quantità R_0 è chiara anche ai fini del controllo. Se avessimo a disposizione un vaccino (perfetto) dovremmo vaccinare abbastanza persone da far sı̀ che la quantità R_0u_0 diventi minore di 1. In altre parole, la percentuale minima da vaccinare per eliminare un’infezione è pari a 1 – \frac{1}{R_0 }. Sono state ottenute stime di R_0 per varie infezioni; per esempio le stime di R_0 per il vaiolo erano fra 5 e 7, cosı̀ come quelle per la polio; invece le stime di R_0 per il morbillo variano fra 12 e 18. Questo spiega il target del 95% che ci si pone per la vaccinazione del morbillo, e spiega anche perché sia più facile eradicare il vaiolo (il grande successo delle campagne di vaccinazione) e la polio (si è arrivati molto vicini al successo) che il morbillo.
Conoscere il valore di R_0 è utile per impostare strategie di controllo, anche se non si ha a disposizione di un vaccino e si vuole intervenire tramite la riduzione del tasso di contatto. Se per esempio R_0 fosse uguale a 2, dovremmo più che dimezzare i tassi di contatto per controllare l’epidemia; se fosse uguale a 3, la riduzione dovrebbe essere proporzionalmente maggiore. Per l’infezione da coronavirus, le stime di R_0 fatte sulla base dei dati osservati in Cina3 variano in genere fra 2 e 3; questo può dare un’idea dello sforzo necessario per il suo contenimento.
Ma come si sono ottenute queste stime di R_0? In sostanza si sono trovate inserendo (4) all’interno di (3) e ottenendo (supponendo inoltre u_0 = 1) \label{r_R0} (5)\ \ \ \ \ \ \ R_0 = \frac{ \int_0^\infty \pi(\tau) \phi(\tau) \, d\tau}{ \int_0^\infty \pi(\tau) \phi(\tau) e^{-r \tau} \, d\tau}. Se conosciamo le funzioni \pi(\tau) e \phi(\tau) (o qualche loro approssimazione) e abbiamo stimato r, ne otteniamo una stima di R_0. Stimare r è in realtà abbastanza semplice se possiamo osservare la curva dei nuovi casi i(t); supponendo, come detto sopra, che all’inizio dell’epidemia si abbia i(t) \approx C e^{rt}, possiamo stimare r con una regressione lineare di \log(i(t)) su t. Un vantaggio di questo metodo è che funziona anche se non vengono riportati tutti i casi ma solo una frazione p (per esempio quelli ospedalizzati perché più gravi); purché il fattore di sottonotifica p rimanga costante nel tempo, la stima di r rimane non distorta.
Metodi più elaborati per stimare i parametri epidemiologici si basano sull’analisi delle sequenze geniche dei virus e la ricostruzione degli alberi di trasmissione; si è sviluppata una branca statistica, nota come filodinamica4, dedicata a questi problemi, ma ciò esula dal livello di questo articolo.
Parlando di stime dei parametri epidemiologici all’inizio di un’epidemia, vale la pena chiarire perché è difficile ottenere una buona stima di una quantità che è certamente molto importante, come la letalità di un’infezione. Si potrebbe pensare che basti calcolare il rapporto fra il numero dei morti e quello dei casi registrati a un certo tempo t per avere una stima della probabilità di morire a causa dell’infezione. Al di là del fatto che la mortalità dipenderà dalle condizioni dell’infettato, e in particolare dalla sua età, la stima della mortalità tramite il rapporto fra il numero dei morti e quello dei casi registrati soffre di due problemi. Prima di tutto, mentre il numero dei morti è in genere abbastanza certo, i casi registrati sono in genere solo una parte delle infezioni effettivamente avvenute. Il secondo problema è che in genere il tempo di degenza ospedaliera fino alla morte può essere più o meno lungo; quindi al tempo t possiamo conoscere chi è morto al tempo t, ma non sappiamo quanti fra i casi già registrati al tempo t moriranno per la malattia nei prossimi giorni o settimane. Il primo fattore ci fa sovrastimare la mortalità, il secondo, specialmente in un periodo di crescita esponenziale dei casi, ce la fa sottostimare. Quando ci sono tanti dati disponibili, è possibile effettuare correzioni adeguate; in una prima fase, invece, le stime della mortalità non sono molto attendibili.
Il modello SIR con equazioni differenziali ordinarie
La formula che collega R_0 e r diventa molto più semplice, se supponiamo che l’infettività \phi sia costante e che il periodo di infezione segua una distribuzione esponenziale \pi(\tau) = e^{-\gamma \tau}. Allora (5) diventa \label{r_R0_exp} R_0 = 1 + \frac{r }{\gamma } = 1 + r T_I dove T_I rappresenta la lunghezza media del periodo di infettività. Questa formula si usa spesso come approssimazione anche nel caso in cui la distribuzione del periodo di infettività non sia esponenziale, anche, se come visto sopra, ciò non è corretto. Relazioni esplicite si possono ottenere anche nel caso si scelgano distribuzioni diverse dall’esponenziale.
L’ipotesi che il periodo di infettività segua la distribuzione esponenziale, equivale a supporre che la probabilità di guarire non dipenda da quanto a lungo un individuo è stato infetto; come è intuitivo, in genere le distribuzioni effettive sono molto lontane dall’esponenziale. Però la distribuzione esponenziale ha il grosso vantaggio di trasformare il sistema (1) consistente in un’equazione differenziale e un’equazione integrale accoppiate in un sistema di equazioni differenziali ordinarie che sono molto piu semplici da analizzare e da approssimare al computer. Il sistema che si ottiene è \label{system-ODE} (6)\ \ \ \ \ \ \ \begin{array}{lcl} S'(t) & = & – \beta \phi \frac{S(t)}{N } I(t) \\ I'(t) & = & \beta \phi \frac{S(t)}{N } I(t) – \gamma I(t). \end{array} dove I(t) = \int_0^\infty i(t-\tau) e ^{-\gamma \tau}\, d\tau rappresenta il numero totale di infetti. Questo modello è noto come il modello SIR, perché gli individui partono tutti dallo stato S, una parte di loro entra in I e infine in R.
Volendo andare un po’ più vicino alla realtà, pur rimanendo nell’ambito delle equazioni differenziali ordinarie, si può introdurre una classe intermedia, E, di individui che sono stati infettati ma non sono ancora infettivi, ottenendo il modello SEIR. Il periodo che gli individui passono nello stadio E si può considerare come il periodo di incubazione, anche se nella realtà è possibile che gli individui infetti siano in grado di trasmettere l’infezione prima di manifestare sintomi.
Come abbiamo detto, le soluzioni del sistema (6), cosı̀ come quelle del sistema (1) hanno una prima fase di crescita esponenziale a tasso r, positivo purché R_0 u_0 > 1, come si suppone in genere (altrimenti non si osserverebbe l’epidemia). Anche modelli più complessi, in cui, come accennato, la popolazione viene stratificata in base a molte altre caratteristiche, hanno questa proprietà e le relazioni fra r e R_0 sono simili a quanto visto per la formula (5).
Nel caso di modelli in cui la popolazione è strutturata in gruppi con diverse caratteristiche, un metodo generale per la definizione ed il calcolo di R_0 è stato proposto da Diekmann et al.5.
Il tasso finale di attacco di un’epidemia
Chiaramente la fase di crescita esponenziale non può andare avanti per sempre, anche qualora non vengano messe in atto strategie di controllo o mitigazione. Infatti il numero di suscettibili S(t) non potrà che diminuire (non sono previsti ingressi di nuovi suscettibili) e a un certo punto si arriverà a R_0 \frac{S(t)}{ N} = 1; a quel punto, si sarà raggiunto il picco di infetti, e l’epidemia comincierà a declinare, finché (in un tempo infinito, secondo il modello matematico in cui il numero di infetti può raggiungere frazioni minime, ma non può mai diventare uguale a 0) non ci saranno più infetti, mentre sarà rimasta una certa frazione ancora suscettibile. La frazione rimasta suscettibile alla fine dell’epidemia secondo i modelli (6) o (1) si può calcolare esattamente, e dipende solo da R_0; nel pannello di destra della Figura 1 qui sotto, si mostra come il tasso di attacco (ossia la frazione della popolazione che è stata infettata) dipenda da R_0, supponendo che non vi sia immunità iniziale.
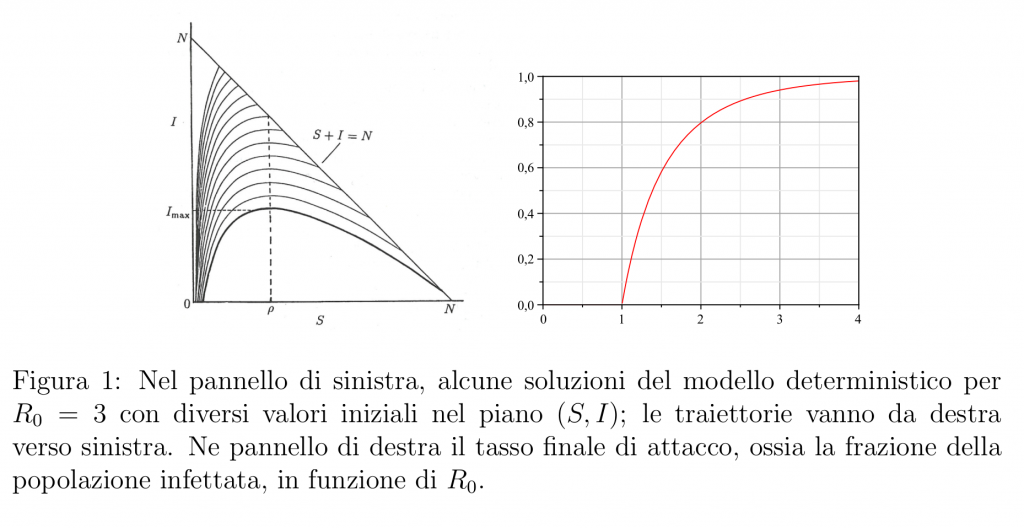
Si vede come già per R_0 = 2 il modello preveda che circa l’80% della popolazione venga infettata nel corso di un’epidemia.
Però la previsione ottenuta tramite questo modello è quasi certamente una sovrastima di quanto ci si possa aspettare in realtà. Prima di tutto, modelli più complessi in cui si tiene conto dell’eterogeneità della popolazione prevedono quasi sempre, a parità di R_0 o di r, un tasso di attacco più basso di quello del modello con “mixing” omogeneo, anche se questa proprietà si riesce a dimostrare solo per alcune classi di modelli. Inoltre, al di là di eventuali strategie di prevenzione imposte o suggerite dalle autorità, è probabile che, proprio a causa della paura indotta da un’infezione poco conosciuta, cambino i comportamenti individuali, riducendo i contatti. Una parte della teoria delle epidemie, la cosiddetta epidemia comportamentale, cerca di modellizzare questo tipo di fenomeni; mi sembra difficile che questi modelli riescano ad essere predittivi, ma certamente comportamenti di auto-prevenzione tendono a ridurre la velocita di diffusione di un’epidemia e anche il suo tasso di attacco.
Il cenno ai comportamenti individuali è l’occasione per ricordare che il valore di R_0 in parte dipende dalla biologia del virus e delle sue interazioni col sistema immunitario che ne determinano l’infettività e la durata del periodo infettivo, ma in parte dipende dalla struttura dei contatti fra gli individui. Mentre le stime ottenute in Cina sul periodo di incubazione e sull’intervallo seriale dovrebbero essere approssimativamente valide dovunque, non è detto che il valore di R_0 debba essere uguale in Cina, in Italia o in Nuova Zelanda.
La dinamica dell’epidemia da coronavirus in Cina in cui da inizio febbraio vi è una discesa costante del numero giornaliero di nuovi casi accertati (vedi Figura 2) mostra che misure draconiane di isolamento sono effettivamente in grado di controllare un’epidemia. La domanda naturale che sorge è quanto a lungo sia necessario mantenerle in vigore.
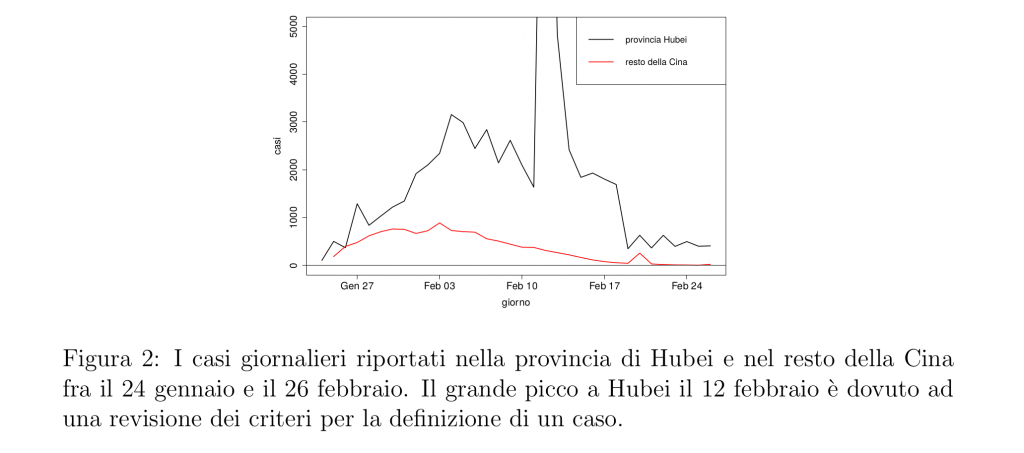
Se usiamo il modello SIR dato dal sistema (6) o comunque qualsiasi modello deterministico in cui il numero di infetti sia descritto tramite una variabile continua, la risposta è “per sempre”: nel modello, se sospendiamo le misure al tempo T quando il numero di infetti previsto I(T) è uguale, per esempio, a 10^{-20}, l’epidemia riparte dopo il tempo T, raggiungendo un picco simile a quello che si sarebbe ottenuto inizialmente senza misure di controllo.
È chiaro che in questo caso il modello sta fornendo previsioni insensate; in realtà quando l’ultimo infetto sarà guarito, il virus sarà eradicato e l’epidemia non potrà ripartire, cosı̀ come è avvenuto per la SARS nel 2003. Per studiare dal punto di vista matematico i fenomeni di estinzione (dell’epidemia o di popolazioni in genere) è necessario passare allora ad un modello stocastico, in cui il numero di infetti viene descritto non tramite una variabile continua, ma da un numero intero, ed in cui le transizioni (infezioni, guarigioni,…) fra stati avvengono in modo casuale. È possibile ottenere risultati pressoché completi anche per un modello stocastico equivalente al sistema (1) che ammette una distribuzione arbitraria del periodo di infezione ed infettività variabile nel corso di esso.
Conclusioni
Finora abbiamo considerato un’epidemia su una scala breve di tempo, ignorando nascite e morti. L’epidemia si estingue perché non ci sono più abbastanza suscettibili, ossia R_0 \frac{S(t)}{N } diventa minore di 1 per t grande. Cosa succede se consideriamo le nascite (oltre alle morti) che porteranno nuovi suscettibili nel sistema?
Nei modelli deterministici la risposta è abbastanza semplice: l’infezione diventerà endemica, con le frazioni suscettibili e immuni che, nei modelli più semplici, tenderanno a un equilibrio; in modelli più complessi, potrà anche succedere, a seconda delle ipotesi fatte, che le frazioni suscettibili e infette abbiano oscillazioni regolari o anche irregolari (caotiche) in alcuni casi. Se si usano modelli stocastici, che non possiamo trattare in questa sede, la risposta è meno chiara ed é ancora oggetto di studio.
Concludendo, ho cercato di fare un breve riassunto di alcuni concetti che emergono dalla modellizzazione matematica delle epidemie e di come possono aiutare anche nel comprendere la situazione nel caso di nuove infezioni e nel formulare strategie di controllo adeguate. Ho anche cercato di mostrare alcuni dei problemi matematici che nascono da questi modelli. Esistono infatti ancora fenomeni non completamente compresi, specialmente nel caso di modelli stocastici.
Andrea Pugliese
Dipartimento di Matematica
Università di Trento
- ripubblicato in Bull.Math.Biol.53:33-55 (1991)↩
- New England J. Med., https://www.nejm.org/doi/full/10.1056/NEJMoa2001316↩
- Wu et al., The Lancet, https://doi.org/10.1016/S0140-6736(20)30260-9↩
- Rasmussen et al., PLoS Comput Biol, 10(4):e1003570↩
- Diekmann et al. J.Math.Biol.28:365-382 (1990)↩





![Giornata Internazionale delle Donne nella Matematica 2024 – Tutti gli eventi italiani [AGGIORNATO] may12_2024](https://maddmaths.simai.eu/wp-content/uploads/2024/04/may12_2024-300x188.png)












Articolo confuso e impreciso:
– formule e passaggi matematici non sono spiegati o lo sono solo sommariamente
– la figura 1 non è spiegata quasi per niente e non si chiarisce come sono ricavate le curve nè il tasso di attacco
– mortalità e letalità sono usate come se fossero sinonimi
– R0 viene considerato funzione della % di suscettibili mentre è fisso, quello che varia è Rt che non è mai citato
– è insensato prendere il numero degli infetti previsti pari a 10^-20 della popolazione mondiale: non può essere minore di 1,3 * 10^-10 (1 individuo su 7,7 mld di abitanti)
Sarebbe il caso di aggiornarlo ed emendarlo
Ringrazio per l’attenzione e mi spiace di avere deluso le sue aspettative; certamente la mancanza di chiarezza è colpa mia.
Mi era parso che in questa sede non fosse opportuno dare i dettagli dei passaggi matematici necessari per raggiungere i risultati, ma fosse meglio concentrare l’attenzione su quelli che ritenevo i concetti più importanti.
– Per esempio, la curva mostrata nel pannello di destra della Figura 1 si trova risolvendo un’equazione trascendente, ottenuta tramite un’analisi del sistema (6). I dettaglli si trovano su molti libri, e sull’articolo originale di Kermack e McKendrick.
– R0, come presentato in (4), non è funzione della % di suscettibili. Mi scuso se in altri punti del testo ho dato l’impressione che lo sia. R_t è essenzialmente un concetto statistico, più che modellistico. Si cerca di stimare quale sia, al tempo t, il numero medio effettivo di individui infettato da ogni infetto; quindi, esso dipende sia della riduzione della frazione suscettibile, sia dai cambiamenti di comportamenti che influiscono sui tasso di contatto. Data l’attenzione che viene data a questa stima, sicuramente se avessi scritto questo articolo ultimamente, avrei dedicato un po’ di spazio a R_t, anche se appunto si tratta di un metodo per descrivere lo stato attuale della dinamica epidemica più che per prevederne l’andamento futuro.
– Sull’ultimo punto, evidentemente non sono stato capace di farmi capire. Era un esempio per far capire che le previsioni dei modelli (in particolare di quelli in cui la popolazione viene descritta come una variabile continua) non vanno prese alla lettera, ma possono essere insensate, come quando si prevederebbe che la frazione di infetti diventi 10^{-20} e poi aumenti di nuovo.
Spiegazione precisa e accurata. Ulteriori chiarimenti su Ro ed Rt a carattere più divulgativo sono forniti da questo articolo al link
Salve
Ma può verificarsi che con R0 <1 il numero delle persone infette aumenti,( nonostante con R0<1 la tendenza sia verso la risoluzione)?
Ps sono un medico non un matematico, scusate l'ignoranza in tal campo
ho trovato interessante anche quanto pubblicato a questo link
https://www.academia.edu/42393382/Sviluppo_di_un_modello_alle_differenze_finite_per_la_simulazione_di_epidemie
hanno pubblicato una versione decisamente più aggiornata di questi appunti il link è :
https://www.academia.edu/42556114/Note_sullo_sviluppo_di_un_modello_alle_differenze_finite_per_la_simulazione_di_epidemie_AGGIORNAMENTO2
anche io lo sto trovando un approccio interessante
…un classico modo di affrontare il problema da ingegnere…
Fino ad un mese fa gli avrei consigliato di studiare di più.
Oggi penso che sapere andare al sodo può fare la differenza.
Sicuramente un buon punto di partenza
Ho trovato lo stesso lavoro su zenodo che non richiede registrazione.
https://doi.org/10.5281/zenodo.3756687
grazie , molto utile !
Il parametro latenza del potenziale infetto non è stato parametrizzato. Potrebbe essere di circa 2 mesi. Si perché conosco un soggetto che ha estrinseca la malattia ad metà aprile ed è stato contaminato il 1 febbraio. La latenza è un parametro che solo ora percepiamo…
E anche a questo link
https://clubcdt.it/it/34_T#page
La dI/I (la percentuale di incremento di casi positivi, p.es. quotidiana), è è una grandezza facilmente comprensibile ai più, che intuitivamente collego alla costante r, per lo meno nella fase di crescita esponeziale, ma non sono riuscito a trovarne una derivazione. E’ corretta la mia intuizione? Grazie in ogni caso per la chiara e esaustiva descrizione dei modelli.
A mio avviso la variazione percentuale giornaliera, ancorché “comprensibile ai più”, non è un dato rappresentativo per una serie di motivi: – la base di calcolo varia giornalmente, aumentando costantemente, per cui il confronto tra due o più percentuali consecutive è di per sé disomogeneo e quindi fuorviante; – una percentuale maggiore di zero, alta o bassa che sia, significa solo che il contagio progredisce. Una eventuale tendenza in costante diminuzione della suddetta percentuale, potrebbe forse essere l’unico dato interessante. Però mi piacerebbe sentire il parere di Andrea Pugliese. Saluti!
Riuscirebbe ad ipotizzare quando potrebbe esserci il picco per l’infezione Covid 19 in Italia? O in Lombardia oppure nel Veneto?
Penso che sia più utile la teoria delle reti col concetto di piccoli mondo. Oppure. Osservare con i frattali le varie evoluzioni del fenomeno con piccole variazioni delle costanti. Infine avvisare il virus di adottare il comportamento previsto